

Alle Kurse wurden bereits erfolgreich durchgeführt. Wir haben die Inhalte der Veranstaltung und unsere Erfahrungen mit verschiedenen didaktischen Methoden zusammengefasst und veröffentlicht (Wolff et al., 2023). Interessierte können dort mehr über die sechs Durchgänge dieses Kurses erfahren. Interessiserte Studierende können sich an Boas Pucker oder Katharina Wolff wenden. Wir arbeiten zur Zeit an der Implementierung eines dauerhaften Moduls, um auch weiterhin Studierenden 'Data Literacy' zu vermitteln.
Dieses Projekt stellt eine Lehrinnovation dar, die durch die Stiftung Innovation in der Hochschullehre im Rahmen von Freiraum2022 gefördert wurde.
Kursinhalt und Konzept:
Am Beispiel einer Pflanzengenomsequenzierung sollen Arbeitsschritte eines realen Projektes von den Studierenden geplant und praktisch durchgeführt werden. Hierbei liegt ein Fokus auf der integrierten Vermittlung von Inhalten unterschiedlicher Fächer einschließlich Biochemie, Molekularbiologie, Genomik und Bioinformatik. Das Projekt beginnt mit der Beachtung rechtlicher Vorgaben wie dem Nagoya-Protokoll bei der Probenauswahl. Außerdem lernen die Studierenden Informationen aus diversen Quellen zusammenführen, um ihr Projekt optimal zu planen. Anschließend werden sie ihre Arbeitsschritte im Labor planen, um hochmolekulare DNA zu extrahieren und eine Sequenzierung per Nanopore-Technologie (MinION) durchzuführen. Eine besondere Herausforderung besteht dabei in der effizienten Ausnutzung der Sequenzierkapazitäten. Die Studierenden übernehmen hierbei Verantwortung für wertvolle Geräte und Materialien. Molekularbiologische Labore und Geräte werden von meiner Gruppe zur Verfügung gestellt. Alle Arbeitsschritte werden von den Studierenden in einem digitalen Laborbuch dokumentiert. Dabei sollen die Studierenden gegenseitig ihre Dokumentation überprüfen und Vorschläge für Verbesserungen machen (peer-review). Damit wird das Verständnis für wichtige Details in den Metadaten durch kooperatives Lernen geschärft. Mit digitalen Laborbüchern und peer-review zur Protokoll-Kontrolle habe ich bereits in einem vom Stifterverband geförderten Projekt gute Erfahrungen gemacht. Ein weiteres innovatives Element dieses Projekts ist die Präsentation des Fortschritts über social media. Dadurch sollen erste Kompetenzen für Wissenschaftskommunikation erworben werden. Die geplante Sequenzierung wird große Datensätze generieren, sodass die Studierenden Erfahrung in der Handhabung großer Datensätze sammeln. Diese Datensätze sollen prozessiert werden, wobei eine exakte Dokumentation aller Schritte essentiell ist. Für die Analysen soll eine Cloud-basierte Lösung verwendet werden, um aktuelle Entwicklungen in der Bioinformatik aufzuzeigen. Die Studierenden lernen verlässliche Backuplösungen zu finden, um einen Verlust der Datensätze zu vermeiden. Schließlich sollen die finalen Datensätze über die etablierten Repositorien veröffentlicht werden. Dies beinhaltet das European Nucleotide Archive für die Sequenzierdaten. Dabei ist eine genaue Beschreibung des Sequenzierprozesses notwendig. In diesem Kontext sollen die Studierenden das Konzept von FAIR (Findable Accessible Interoperable Reuseable) data verinnerlichen. Verschiedene Optionen zur Veröffentlichung von Datensätzen sollen im Hinblick auf ihre Vor- und Nachteile von den Studierenden verglichen werden. Als Abschluss der Veranstaltung sollen die Studierenden ihre Ergebnisse vor einem internationalen Publikum im Rahmen einer selbstorganisierten Online-Konferenz präsentieren.
Lernziele:
Studierende sollen befähigt werden selbstständig datenintensive Projekte zu planen, durchzuführen und vollständig zu dokumentieren (data literacy); Genomsequenzierungsprojekte und andere omics-Projekte selbständig planen und durchführen können; große Datensätze Cloud-basiert analysieren und ihre Ergebnisse veröffentlichen können; ihre Arbeit und deren Ergebnisse gegenüber einer interessierten Öffentlichkeit und und einem Fachpublikum präsentieren können.
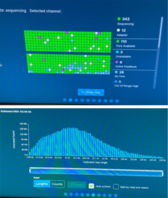
Einige Eindrücke der ersten Woche des ersten "Data Literacy in Genome Research"-Kurses.

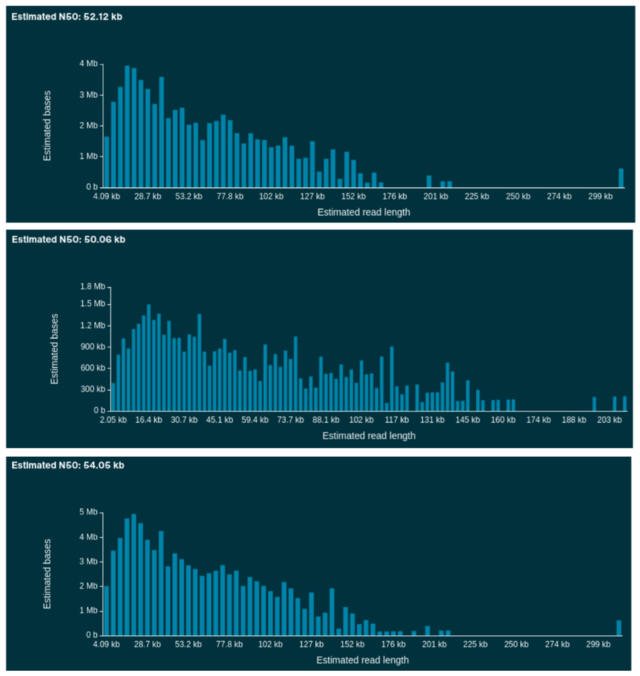





Die Folien der Vorlesung sind über GitHub (Data Literacy in Genome Research) frei verfügbar. Eine Verwendung in anderen Lehveranstaltungen ist (möglichst unter Angabe der Quelle) ausdrücklich gestattet. Unsere Protokolle zur Durchführung der praktischen Experimente sind über protocols.io ebenfalls frei zugänglich: "Plant DNA extraction and preparation for ONT sequencing".
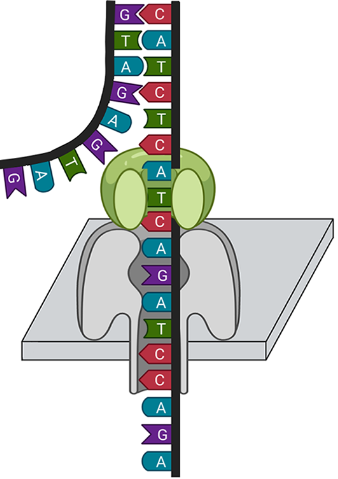
Wir haben Details zur Technologie, zum aktuellen Stand des Forschungsfelds und zur zukünftigen Entwicklung in einem Review-Artikel beschrieben: "Plant genome sequence assembly in the era of long reads: Progress, challenges and future directions"